Ya comentaré con más detenimiento el código, pero es la unión de muchos de los códigos R de días anteriores, es un buen ejemplo de uso de la librería gridExtra para poner múltiples gráficos en una sola salida:
library(dplyr)
library(ggplot2)
library(reshape)
library(gridExtra)
df <- read.csv("https://raw.githubusercontent.com/datadista/datasets/master/COVID%2019/ccaa_covid19_fallecidos.csv",
sep=',', check.names=FALSE, encoding = 'UTF-8')
df2 <- melt(df[,-1])
names(df2) = c('CCAA','fecha','fallecidos')
mm <- df2 %>% group_by(CCAA) %>% summarise(total_fallecidos = sum(fallecidos)) %>% arrange(desc(total_fallecidos)) %>%
mutate(CCAA2 = ifelse(row_number()>=10,'Resto', as.character(CCAA))) %>% select(CCAA,CCAA2)
df2 <- left_join(df2,mm)
table(mm$CCAA2)
df2 <- df2 %>% group_by(CCAA2,fecha) %>% summarise(fallecidos=sum(fallecidos)) %>%
mutate(fecha = as.Date(as.character(fecha),origin='1970-01-01')) %>% as_tibble()
df3 <- df2 %>% mutate(fecha=fecha+1, fallecidos_anterior=fallecidos) %>% select(-fallecidos)
df2 <- left_join(df2, df3) %>% mutate(fallecidos_dia = fallecidos - fallecidos_anterior)
#Función para hacer los gráficos
grafica <- function(comunidad){
p <- ggplot(filter(df2,CCAA2==comunidad), aes(x=fecha)) +
geom_line(aes(y=fallecidos_dia, group = 1), alpha = 0.5, color='red') +
geom_smooth(aes(y=fallecidos_dia), method = "loess") +
ggtitle(comunidad) +
xlab("") + ylab("Fallecidos por día")
return(p)}
madrid = grafica('Madrid')
cat = grafica('Cataluña')
mancha = grafica('Castilla-La Mancha')
leon = grafica('Castilla y León')
pvasco = grafica('País Vasco')
valencia = grafica('C. Valenciana')
andalucia = grafica('Andalucía')
aragon=grafica('Aragón')
resto = grafica('Resto')
total = grafica('Total')
grid.arrange(madrid, cat, mancha, leon, pvasco, valencia, andalucia, aragon, resto, total, nrow=5,ncol=2)
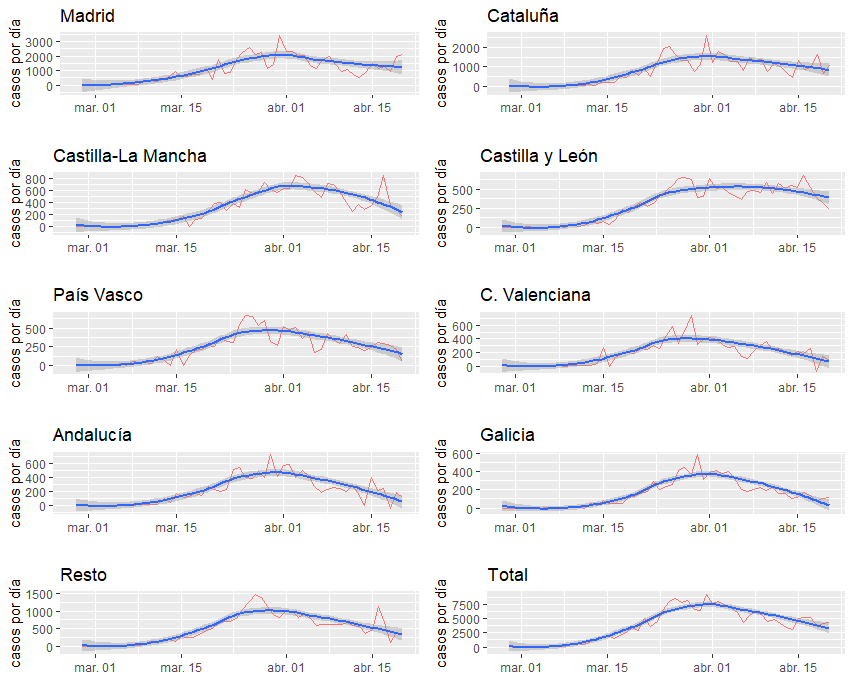
Del mismo modo podemos hacer el número de casos:
# Casos
df <- read.csv("https://raw.githubusercontent.com/datadista/datasets/master/COVID%2019/ccaa_covid19_casos.csv",
sep=',', check.names=FALSE, encoding = 'UTF-8')
df2 <- melt(df[,-1])
names(df2) = c('CCAA','fecha','casos')
mm <- df2 %>% group_by(CCAA) %>% summarise(total_casos = sum(casos)) %>% arrange(desc(total_casos)) %>%
mutate(CCAA2 = ifelse(row_number()>=10,'Resto', as.character(CCAA))) %>% select(CCAA,CCAA2)
table(mm$CCAA2)
df2 <- left_join(df2,mm)
df2 <- df2 %>% group_by(CCAA2,fecha) %>% summarise(casos=sum(casos)) %>%
mutate(fecha = as.Date(as.character(fecha),origin='1970-01-01')) %>% as_tibble()
df3 <- df2 %>% mutate(fecha=fecha+1, casos_anterior=casos) %>% select(-casos)
df2 <- left_join(df2, df3) %>% mutate(casos_dia = casos - casos_anterior)
#Función para hacer los gráficos
grafica <- function(comunidad){
p <- ggplot(filter(df2,CCAA2==comunidad), aes(x=fecha)) +
geom_line(aes(y=casos_dia, group = 1), alpha = 0.5, color='red') +
geom_smooth(aes(y=casos_dia), method = "loess") +
ggtitle(comunidad) +
xlab("") + ylab("casos por día")
return(p)}
madrid = grafica('Madrid')
cat = grafica('Cataluña')
mancha = grafica('Castilla-La Mancha')
leon = grafica('Castilla y León')
pvasco = grafica('País Vasco')
valencia = grafica('C. Valenciana')
andalucia = grafica('Andalucía')
galicia=grafica('Galicia')
resto = grafica('Resto')
total = grafica('Total')
grid.arrange(madrid, cat, mancha, leon, pvasco, valencia, andalucia, galicia, resto, total, nrow=5,ncol=2)
En este caso cambiamos Aragón por Galicia. También cabe destacar que es un buen ejemplo de uso de melt para transponer columnas a filas, al hacer eso el lag lo realizamos mediante left join sumando un día y así podemos calcular la diferencia diaria con el acumulado, esperemos que este tipo de análisis tan burdos se estén llevando a cabo en otros sitios donde toman decisiones. Saludos.