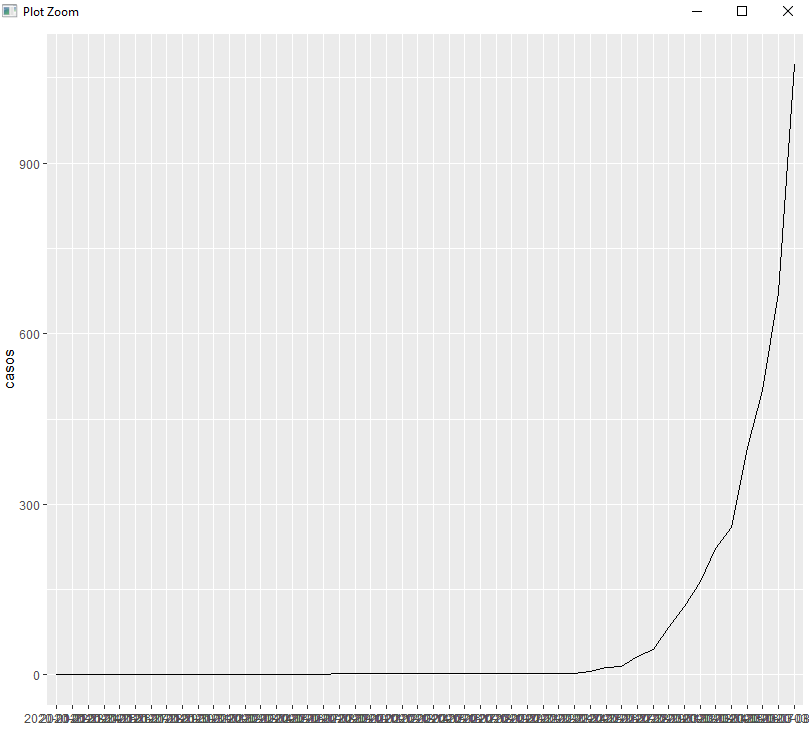
No he podido evitarlo, os traigo unas líneas de código en R para seguir la evolución del coronavirus en España (podéis filtrar cualquier país). Me hubiera gustado hacer un scraping de la página https://www.worldometers.info/coronavirus/ sin embargo me ha parecido más sencillo leer directamente los datos del repositorio de la Universidad Jonh Hopkins (https://github.com/CSSEGISandData/COVID-19) creo que la actualización es diaria. También existe ya un paquete en R denominado coronavirus pero su funcionamiento deja que desear. Por mi parte os ofrezco para seguir su evolución el siguiente script:
library(lubridate)
library(ggplot2)
datos <- read.csv2("https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Confirmed.csv",
sep=',')
fechas <- seq(as.Date("2020/01/22"), as.Date(today()-1), "days")
fechas <- as.character.Date(fechas)
names(datos) <- c("Provincia", "Pais","Latitud", "Longitud", fechas)
espania <- datos %>% filter(Pais=="Spain") %>% select(fechas)
espania <- data.frame(t(espania))
espania$fecha <- row.names(espania)
names(espania) <- c("casos", "fecha")
p <- ggplot(espania, aes(x=fecha, y=casos, group = 1)) +
geom_line() +
xlab("")
p
Tendría que mejorar los ejes y el aspecto, pero no es eso lo más importante. Estaba escribiendo sobre distribuciones tweedie, ahora me siento tentado para escribir sobre modelos exponenciales y si hacéis esto mismo para los datos de Italia hace 10 días la verdad es que el gráfico es calcado.