La regresión no lineal se da cuando tenemos que estimar Y a partir de una función del tipo Y=f(X,Beta) + Error donde Beta son Beta1, Beta2,…, Beta n. Unos datos X e Y se relacionan mediante una función no lineal respecto a unos parámetros Beta desconocidos. Y cómo obtenemos estos Beta desconocidos, a través de mínimos cuadrados o bien con otros métodos como máxima verosilimilitud. Este cálculo llevará asociada su inferencia estadística habitual. La función que asocia los pares de datos (x1,y1), (x2, y2),…, (yn, xn) será una función conocida. Por eso esta técnica es muy utilizada en ciencias químicas, geodinámica,… donde ya se conoce la relación que hay entre las variables independientes y la variable dependiente pero es necesario realizar modelos con los pares de datos disponibles de cara a obtener estimaciones.
Para la realización de este monográfico con R vamos a emplear el conjunto de datos Thurber Son datos de un estudio NIST de movilidad de electrones en semiconductores la variable respuesta es la movilidad del electrón y la variable regresora es el logaritmo de la densidad. El modelo es:
Nuestra variable está relacionada con su regresora por un modelo racional con siete parámetros Beta1, Beta2,…, Beta7 y cúbicas. Comenzamos el trabajo con los datos en R:
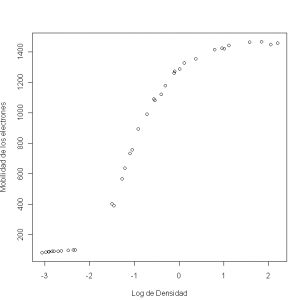
y = c(80.574 ,84.248 ,87.264 ,87.195 ,89.076 ,89.608 ,89.868 , 90.101 ,92.405 ,95.854 ,100.696 ,101.06 ,401.672 ,390.724 , 567.534 ,635.316 ,733.054 ,759.087 ,894.206 ,990.785 ,1090.109 , 1080.914 ,1122.643 ,1178.351 ,1260.531 ,1273.514 ,1288.339 , 1327.543 ,1353.863 ,1414.509 ,1425.208 ,1421.384 ,1442.962 , 1464.35 ,1468.705 ,1447.894 ,1457.628) x = c(-3.067 ,-2.981 ,-2.921 ,-2.912 ,-2.84 ,-2.797 , -2.702 ,-2.699 ,-2.633 ,-2.481 ,-2.363 ,-2.322 ,-1.501 ,-1.46 , -1.274 ,-1.212 ,-1.1 ,-1.046 ,-0.915 ,-0.714 ,-0.566 ,-0.545 , -0.4 ,-0.309 ,-0.109 ,-0.103 ,0.01 ,0.119 ,0.377 ,0.79 ,0.963 , 1.006 ,1.115 ,1.572 ,1.841 ,2.047 ,2.2) #Representación de los datos plot(y ~ x,xlab="Log de Densidad", ylab="Mobilidad de los electrones")
Metemos los datos directamente desde la web a R. Realizamos una representación gráfica de los datos y se aprecia la inexistencia de relación lineal. Se nos indica que existe relación entre las variables mediante la función antes indicada. Introduzcamos en R la función:
foo = function(x,b1,b2,b3,b4,b5,b6,b7){
(b1 + b2*x + b3*x^2 + b4*x^3)/
(1 + b5*x + b6*x^2 + b7*x^3)}
El trabajo con R le vamos a llevar a cabo con la función nls del paquete stats. Pero antes de crear un modelo de regresión no lineal tenemos que asignar unos valores iniciales a los parámetros Beta de nuestra ecuación. La regresión no lineal es un proceso iterativo. Se parte de unos parámetros Beta iniciales, se modeliza y mediante un proceso de optimización numérica se aproximan los parámetros seleccionados a los valores óptimos. Si empleamos el algoritmo de Gauss-Newton partiríamos de la mínima suma de cuadrados de los residuos (modelo lineal) y tomaríamos esta función como función a minimizar algo que es posible debido a que al menos una derivada depende de uno de los parámetros Beta (condición de no linealidad). El proceso busca mínimos locales de la función y que posteriormente habrá de comprobar si son mínimos globales hasta que el proceso llegara a converger (o no). Para obtener los valores iniciales es necesario conocer los datos. En nuestro caso tenemos una división y 7 parámetros. Vamos a observar la gráfica con los datos. Para x=0 el valor de y ha de ser muy próximo a 1200, luego ese es un buen inicio para b1. No podemos tener valores negativos, luego los parámetros que multiplican tanto a x como a x^3 no deberían ser los más altos. Además tenemos una división y luego los parámetros que estén en el denominador no deberían ser muy altos ya que la función ha de ser creciente. Si comenzamos a ejecutar:
plot(y ~ x,xlab="Log de Densidad", ylab="Mobilidad de los electrones",main="Prueba 1") curve(foo(x,1200,100,100,1,0.1,1,0.1),add=T,col="red")
Vemos que hasta 0 la curva no tiene mal aspecto, pero a desde ese punto la forma no es adecuada. Los parámetros del denominador pueden ser más bajos:
plot(y ~ x,xlab="Log de Densidad", ylab="Mobilidad de los electrones",main="Prueba 2") curve(foo(x,1200,100,100,1,-0.1,0.1,-0.1),add=T,col="blue")
Realicemos un primer modelo con estas especificaciones:
m1start=list(b1=1200,b2=100,b3=100,b4=1,b5=-0.1,b6=0.1,b7=-0.1)
Obtenemos el error Error en nlsModel(formula, mf, start, wts) : singular gradient matrix at initial parameter estimates Este error se produce debido a que los valores iniciales no son correctos para poder realizar el algoritmo inicial ya que no es posible encontrar un primer gradiente. Podríamos ir realizando diversas pruebas para encontrar los valores iniciales e incluso elaborar una parrilla de datos. También podemos emplear la librería nls2:
#install.packages("nls2")
library(nls2)
m1<- nls2(y ~ foo(x,b1,b2,b3,b4,b5,b6,b7),
start=c(b1=1200,b2=100,b3=100,b4=1,b5=-0.1,b6=0.1,b7=-0.1),
control = nls.control(warnOnly = TRUE))
Con esta librería nls2 estamos empleando el algoritmo “brute force” que se emplea para encontrar los valores iniciales. Es importante destacar que no se emplea para realizar el modelo, sólo para resolver la problemática de los valores iniciales. En este ejemplo concreto se sabe que los valores iniciales son:
B1=1000 B2=1000 B3=400 B4=40 B5=0.7 B6=0.3 B7=0.03 m.nls = nls(y ~ foo(x,b1,b2,b3,b4,b5,b6,b7), start = c(b1 = 1000, b2 = 1000, b3 = 400, b4 = 40, b5 = 0.7, b6 = 0.3, b7 = 0.03),trace=T)
En este caso ya hemos obtenido resultados. Con summary(m.nls) la salida obtenida es:
Formula: y ~ foo(x, b1, b2, b3, b4, b5, b6, b7)
Parameters:
Estimate Std. Error t value Pr(>|t|)
b1 1.288e+03 4.665e+00 276.141 < 2e-16 ***
b2 1.491e+03 3.957e+01 37.682 < 2e-16 ***
b3 5.832e+02 2.870e+01 20.323 < 2e-16 ***
b4 7.542e+01 5.567e+00 13.546 2.55e-14 ***
b5 9.663e-01 3.133e-02 30.840 < 2e-16 ***
b6 3.980e-01 1.499e-02 26.559 < 2e-16 ***
b7 4.973e-02 6.584e-03 7.553 2.02e-08 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 13.71 on 30 degrees of freedom
Number of iterations to convergence: 28
Achieved convergence tolerance: 8.36e-06
El algoritmo ha necesitado de 28 iteracciones, vemos los estimadores de los parámetros. Ahora es necesario que realicemos un diagnóstico del modelo. Comenzamos por graficar el resultado del modelo:
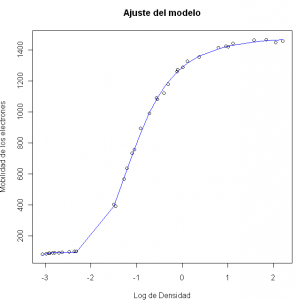
plot(y ~ x,xlab="Log de Densidad", ylab="Mobilidad de los electrones",main="Ajuste del modelo") lines(x,fitted(m.nls),col="blue")
Gráficamente el modelo ajusta bien. Podemos ver la suma del cuadrado de los errores con la función deviance:
deviance(m.nls) [1] 5642.708
También es necesario analizar si el modelo cumple las hipótesis de ser estadísticamente válido con el test F, homocedasticidad, distribución normal de los errores y errores independientes. El test F lo podemos realizar con un test ANOVA con el ajuste por mínimos cuadrados frente a nuestro modelo de regresión no lineal. El código R que podemos emplear para realizar estas tareas es:
#Anova para contraste de falta de ajuste m.lm <- lm(y~x) anova(m.nls,m.lm) #Independencia de los residuos plot(fitted(m.nls),residuals(m.nls), xlab="Valores ajustados",ylab="Residuos") abline(a=0,b=0,col="blue") #Test de normalidad de los residuos qqnorm(residuals(m.nls)) qqline(residuals(m.nls)) shapiro.test(residuals(m.nls)) #Test de Leneve library(car) levene.test(y,as.factor(x)) #Intervalos de confianza confint(m.nls)
No entramos en más detalles para no alargar la entrada. Pero ya disponemos de las herramientas de R para comenzar a trabajar con este tipo de modelos. También recomiendo ver las posibilidades de la librería nlstools. Saludos.
Si el número de iteracciones es mayor a 50 R no ejecuta los parámetros, ¿hay alguna forma de corregir eso?
Hola David,
Puede que sea tarde…pero a alguien le servira
Si quieres incrementar el numero de iteracciones es facil. dentro de la funcion nls tiene la opcion control() que puede modificar algunos parametros (ver ayuda en R). Por defecto nls tiene 50, si quieres aumentarlas a 100 por ejemplo, prueba:
nls(x,y,…,control(maxiter=100))
Good luck!
Hola, y si quiero ajustar los datos a una curva sigmoidal como lo realizaria
hola soy nuevo usando los modelos nls me sale el error Error in nls(PM2.5 ~ A + B * exp(-K * t), data = dat.nls, start = list(A = 51.611, :
singular gradient
mi base de datos es
dat.nls
fecha PM2.5 t
1 10/3/2018 12:52 53 0
2 10/3/2018 12:53 58 1
3 10/3/2018 12:54 81 2
4 10/3/2018 12:55 104 3
5 10/3/2018 12:56 94 4
6 10/3/2018 12:57 95 5
7 10/3/2018 12:58 107 6
8 10/3/2018 12:59 113 7
9 10/3/2018 13:00 146 8
10 10/3/2018 13:01 115 9
11 10/3/2018 13:02 150 10
12 10/3/2018 13:03 159 11
13 10/3/2018 13:04 146 12
14 10/3/2018 13:05 93 13
15 10/3/2018 13:06 132 14
16 10/3/2018 13:07 143 15
17 10/3/2018 13:08 174 16
18 10/3/2018 13:09 173 17
19 10/3/2018 13:10 210 18
20 10/3/2018 13:11 139 19
21 10/3/2018 13:12 129 20
22 10/3/2018 13:13 143 21
23 10/3/2018 13:14 125 22
24 10/3/2018 13:15 122 23
25 10/3/2018 13:16 161 24
26 10/3/2018 13:17 147 25
27 10/3/2018 13:18 156 26
28 10/3/2018 13:19 117 27
29 10/3/2018 13:20 155 28
30 10/3/2018 13:21 173 29
31 10/3/2018 13:22 116 30
32 10/3/2018 13:23 153 31
33 10/3/2018 13:24 165 32
no se que esta mal
Hola, hay una lista de usuarios de R en español. http://r-es.org/2016/03/07/la-lista-de-correo-r-help-es/ Concreta bien tu pregunta con el código y te lo resuelven en 10 minutos. Saludos.
Hola,
Estoy intentando ajustar una exponencial con cosas extra por ahí, utilizo este código:
«Iosc.nls=nls(I ~ Iosc(V,I0,eta,Rsh),start=oscStart2)»
Con los parámetros obtenidos del nls2. Pero me sigue saliendo el siguiete error:
«Error in nlsModel(formula, mf, start, wts) :
singular gradient matrix at initial parameter estimates
Además: Warning message:
In (function (formula, data = parent.frame(), start, control = nls.control(), :
singular gradient»
No sé qué hago mal…
Parece que pusieras algo redundante, pero la fórmula (aparentemente) está bien. ¿Has investigado en stackoverflow?
Hola, excelente tu aporte. Soy nuevo en esto, quisiera saber como puedo encontrar el r, r^2 y el r^2 ajustado a este ejercicio.Alguien podría colaborarme por favor?
Muchas gracias por la información… me sera muy util.