Además de manejar los datos de un data frame en ocasiones es necesario realizar uniones entre conjuntos de datos para crear o añadir nuevas variables a un data frame que es una base de observaciones inicial. Se pueden establecer 2 tipos de uniones fundamentales, uniones verticales de tablas y uniones horizontales. Las uniones verticales serán las concatenaciones de data frames, poner una estructura de datos encima de otra y las uniones horizontales serán las que se denominarán join.
Se emplea una estructura de datos sencilla para ejemplificar el funcionamiento.
library(kableExtra)
library(tidyverse)
df1 <- data.frame(anio = c(2018, 2019, 2020, 2021), variable1=c(10, 20, 30, 40), variable2=c(1000,2000,3000,4000))
df2 <- data.frame(anio = c(2017, 2018, 2019, 2020), variable1=c(50, 60, 70, 80), variable3=c(5000,6000,7000,8000))
df1 %>% kable()
df2 %>% kable()
Se puede observar como se han creado manualmente 2 data frames con lo que trabajaremos y el uso de tidyverse y kable para la visualización de tablas en R. Veamos los principales tipos de uniones.
Uniones verticales
El siguiente código emplea la función rbind.data.frame para concatenar datos, para poner una tabla encima de otra y generaría un error:
df <- rbind.data.frame(df1, df2)
Error in match.names(clabs, names(xi)) : names do not match previous names
Significa que ambos conjuntos de datos no tienen las mismas variables.
En las uniones verticales con la función rbind.data.frame se han de unir las mismas estructuras. En los datos de trabajo no se dispone de la misma estructura por lo que se torna necesario saber que deseamos unir verticalmente, saber que deseamos concatenar. Si deseamos realizar una unión de todos los datos ambas tablas requieren de las mismas variables:
df1variable3 <- NA
df2variable2 <- NA
df <- rbind.data.frame(df1, df2)
df %>% kable()
Se han creado las variables 3 y 2 donde ha sido necesario y ya se está en disposición de concatenar ambos data frames. Observemos como queda el data frame resultante. Es importante puntualizar que se están produciendo duplicidades por la variable anio, cabe preguntarse ¿son necesarias esas duplicidades? Cuando se trabaje con datos es muy importante disponer de un campo identificativo del registro y determinar si existen duplicidades por ese campo.
En cualquier caso, con el paquete dplyr se pueden concatenar data frames mediante la función bind_rows.
df <- bind_rows(df1, df2)
df %>% kable()
El empleo de esta función no es sensible a la necesidad de que ambos conjuntos de datos tengan los mismos nombres de las variables, si eso no ocurre se emplean valores perdidos representados en R como NA para aquellas ocasiones en las que no coincida.
Uniones horizontales o join
Esta conocida figura recoge en SQL todos los tipos de join:
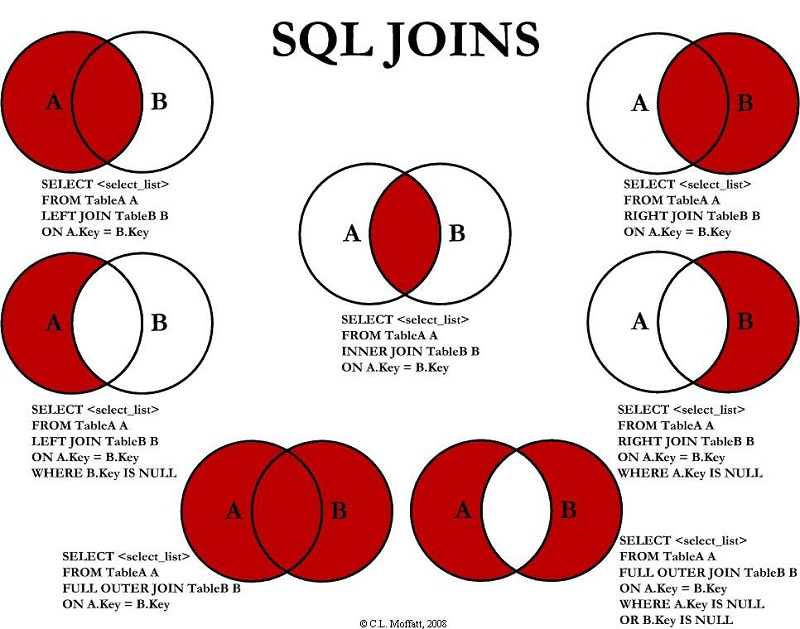
No se considera ver todos los ejemplos, se estudiarán las uniones más habituales en el trabajo diario.
Inner join
Es la intersección de dos conjuntos de datos. Usamos la función inner_join de dplyr.
df1 <- data.frame(anio = c(2018, 2019, 2020, 2021), variable1=c(10, 20, 30, 40), variable2=c(1000,2000,3000,4000))
df2 <- data.frame(anio = c(2017, 2018, 2019, 2020), variable1=c(50, 60, 70, 80), variable3=c(5000,6000,7000,8000))
df <- inner_join(df1,df2, by='anio')
# Equivale a df <- df1 %>% inner_join(df2, by='anio')
df %>% kable()
La unión de ambas estructuras tiene una variable variable1 en común, dplyr entiende que es necesario preservar las variables del conjunto de datos de la derecha, con el sufijo .x, y las variables del conjunto de datos de la izquierda, con el sufijo .y por este motivo es muy relevante determinar que se quiere unir. En los datos de trabajo podríamos saber cuales de los datos de la izquierda coinciden por año con los de la derecha y unir la variable 3.
df2 <- df2 %>% select(-variable1)
df <- inner_join(df1,df2)
df %>% kable()
Se ha eliminado la variable1 del df2 como paso previo, es la que ambos conjuntos de datos tienen en común, se realiza la unión y en este caso se ha obviado el campo de unión porque dplyr busca la «unión natural», el campo en común que es anio en este caso y no es necesario especificar by= con lo que podemos ahorrar código. En el trabajo diario del científico de datos es necesario realizar múltiples uniones de conjuntos de datos por un camo identificativo (roles de las variables), es buena práctica que este campo identificativo tenga el mismo nombre para todos los conjuntos de datos de trabajo.
Left join
Quizá una de las uniones más habituales en el trabajo diario de un científico de datos. Se parte de un conjunto de datos de base y se le añaden nuevas variables por la derecha respetando las observaciones de la izquierda. La función de dplyr usada es left_join.
df1 <- data.frame(anio = c(2018, 2019, 2020, 2021), variable1=c(10, 20, 30, 40))
df2 <- data.frame(anio = c(2017, 2018, 2019, 2020), variable3=c(5000,6000,7000,8000))
df1 <- df1 %>% left_join(df2)
df1 %>% kable()
Se ha añadido por la derecha la variable3 al df1, añadimos una nueva variable a un data frame de base.
Anti join
Vamos se van a seleccionar aquellos registros de una tabla base que no están en otra tabla de cruce.
df1 <- data.frame(anio = c(2018, 2019, 2020, 2021), variable1=c(10, 20, 30, 40))
df2 <- data.frame(anio = c(2017, 2018, 2019, 2020), variable3=c(5000,6000,7000,8000))
df <- df1 %>% anti_join(df2)
df %>% kable()
Se observa que no se ha unido ninguna variable, solo se ha seleccionado el registro de df1 que no cruza con df2.
Librería sqldf
Como científicos de datos es importante saber SQL como lenguaje de consulta, si sabemos SQL tenemos la librería sqldf para utilizar directamente SQL sobre data frames de R.
library(sqldf)
df1 <- data.frame(anio = c(2018, 2019, 2020, 2021), variable1=c(10, 20, 30, 40), variable2=c(1000,2000,3000,4000))
df2 <- data.frame(anio = c(2017, 2018, 2019, 2020), variable1=c(50, 60, 70, 80), variable3=c(5000,6000,7000,8000))
# Inner Join
df <- sqldf("select a.anio, a.variable1, variable3
from df1 a, df2 b
where a.anio = b.anio")
df %>% kable()
# Left join
df <- sqldf("select a.anio, a.variable1, variable3
from df1 a left join df2 b
on a.anio = b.anio")
df %>% kable()
# Anti Join
df <- sqldf("select * from df1 where anio not in (select anio from df2)")
df %>% kable()
Duplicidades en las uniones de tablas
Otra situación habitual que se va a encontrar el científico de datos es la aparición de registros duplicados, es necesario controlar su existencia porque pueden distorsionar el resultado de un análisis.
df1 <- data.frame(anio = c(2018, 2019, 2020, 2021), variable1=c(10, 20, 30, 40))
df2 <- data.frame(anio = c(2017, 2018, 2019, 2020, 2020), variable3=c(5000,6000,7000,8000, 1000))
df <- df1 %>% left_join(df2)
df %>% kable()
En este burdo ejemplo df2 tiene duplicado el año 2020 por lo que una left join con ese conjunto de datos por ese campo provocará duplicidades. Una forma de controlarlo será contabilizar por el campo identificativo.
df %>% group_by(anio) %>% summarise(registros=n()) %>%
filter(registros>1) %>% kable()
En el capítulo anterior ya se anotó la importancia de establecer mecanismos de control cuando se trabajen con datos, bien sea visualizaciones de datos agrupaciones, tablas de frecuencia o estadísticos básicos que veremos en posteriores capítulos.